Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Manual do usuário
- Tipo
- Manual do usuário

Guia do algoritmo de análise de
imagem uPath PD-L1 (SP263)
para o cancro do pulmão de células
não pequenas


Índice
Introdução 1
Resumo e explicação do algoritmo 2
Utilização prevista 3
Utilização prevista do produto 3
Objetivo do Guia do algoritmo 3
Significado clínico 4
Princípios de teste 5
Limitações 6
Proteção de dados 7
Fluxo de trabalho para utilização do algoritmo de análise de imagem
uPath PD-L1 para o NSCLC 8
Fluxo de trabalho do patologista 9
Análise de imagem de PD-L1 12
Avaliação de PD-L1 15
Avaliação da coloração do algoritmo PD-L1 15
Características de coloração 15
Caraterísticas de desempenho 26
Comparação de métodos 26
Estudos de reprodutibilidade de patologista 27
Estudos de reprodutibilidade de digitalizadores 28
Resolução de problemas 29
Referências 33


Introdução
O Roche uPath enterprise software (uPath enterprise software)
com o algoritmo de análise de imagem uPath PD-L1(SP263)
para o cancro do pulmão de células não pequenas (NSCLC)
(algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC) é um sistema de software concebido para ajudar
na avaliação quantitativa da expressão proteica em secções
histológicas coradas imunohistoquimicamente (IHQ) a partir de
tecidos normais e neoplásicos fixados em formol e embebidos em
parafina (FFPE).
O uPath enterprise software é uma solução integral de software
para patologia digital que permite aos laboratórios de patologia
adquirir, gerir, visualizar, analisar, partilhar e gerar relatórios sobre
imagens digitais de espécimes patológicos. Utilizando o uPath
enterprise software, o patologista pode visualizar imagens digitais
em diversas ampliações, atribuir anotações, efetuar medições na
secção de tecido, realizar análises de imagens e gerar relatórios.
Nota: O algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC é uma metodologia auxiliar assistida por computador
que visa ajudar na aquisição e medição de imagens a partir de
lâminas de microscópio de espécimes de tecido corados IHQ, com
vista a verificar a presença da proteína PD-L1. Para assegurar a
validade das pontuações da análise de imagem, o patologista tem
a responsabilidade de verificar a respetiva conformidade utilizando
controlos apropriados, conforme é especificado na folha de
métodos do VENTANA PD-L1 (SP263) Rabbit Monoclonal Primary
Antibody (disponível em www.ventana.com).
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 1

2 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)
Resumo e explicação do algoritmo
No que diz respeito a aplicações de análise de imagem,
o patologista pode utilizar o uPath enterprise software para
selecionar e contornar uma ou mais regiões de interesse (ROI),
podendo cada ROI ser visualizada em várias ampliações e ser,
seguidamente, analisada pelo algoritmo de análise de imagem
uPath PD-L1(SP263) para o NSCLC. É gerada uma contagem do
número total de células tumorais (TC) e as TC são estratificadas
em função de estarem ou não coradas. O algoritmo de análise
de imagem uPath PD-L1(SP263) para o NSCLC divide as TC
coradas pelo número total de TC (coradas e não coradas) para
gerar a pontuação de positividade de PD-L1 TC numa escala
de 0 a 100%. O algoritmo de análise de imagem uPath PD-L1
(SP263) para o NSCLC pode gerar uma pontuação para uma
determinada ROI, ou uma pontuação agregada para todas as ROI
selecionadas nessa lâmina. Embora o algoritmo detete células
não tumorais no âmbito da análise global, a sobreposição e os
dados de saída apenas apresentam as TC utilizadas no cálculo
da pontuação de positividade de TC. O patologista pode aceitar a
pontuação fornecida pelo algoritmo de análise de imagem uPath
PD-L1(SP263) para o NSCLC, ou pode substituir essa pontuação
por uma pontuação diferente. O patologista tem de rever
cuidadosamente tudo o que foi marcado pelo algoritmo como
sendo TC positivas ou negativas e confirmar se o algoritmo está
correto ou efetuar uma substituição manualmente. O algoritmo
de análise de imagem uPath PD-L1(SP263) para o NSCLC não
faz interpretações independentes dos dados e, por isso, só
deve ser utilizado por um patologista qualificado, em conjunto
com um exame histológico, a informação clínica relevante e os
controlos adequados. Foi concebido e está indicado para auxiliar
o patologista na avaliação da expressão da PD-L1 a 50%.

Utilização prevista
Objetivo do Guia do algoritmo
Este Guia do algoritmo de análise de imagem uPath PD-L1 (SP263)
para o NSCLC (Guia do algoritmo) destina-se a:
• Fornecer informações de referência sobre a utilização prevista
do algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC, bem como sobre os princípios e as limitações do
teste.
• Definir os materiais, meios informáticos, segurança de dados
e requisitos de rede necessários.
• Apresentar instruções passo a passo para a execução do
algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC.
• Disponibilizar imagens fotográficas que ilustrem a forma como
algoritmo uPath PD-L1(SP263) de análise de imagem para o
NSCLC deve ser utilizado.
• Colocar à disposição dos patologistas uma ferramenta que
facilite a utilização do algoritmo de análise de imagem uPath
PD-L1(SP263) para o NSCLC em secções de NSCLC FFPE
coradas com o VENTANA PD-L1 (SP263) Assay.
• Disponibilizar imagens exemplificativas de casos complexos,
de modo a explicar a forma como o algoritmo de análise de
imagem uPath PD-L1(SP263) para o NSCLC pode ser utilizado
na respetiva avaliação.
• Apresentar as caraterísticas de desempenho do algoritmo
de análise de imagem uPath PD-L1(SP263) para o NSCLC.
Utilização prevista do produto
Está previsto que o algoritmo de análise de imagem uPath PD-L1
(SP263) para o NSCLC seja utilizado para auxiliar o patologista na
deteção e medição semiquantitativa da proteína PD-L1 em tecido
de NSCLC fixado em formol e embebido em parafina. Quando
utilizado com o VENTANA PD-L1 (SP263) Assay, está indicado
como auxiliar na identificação dos pacientes com NSCLC que
vão efetuar tratamento com terapêuticas com o valor limite de
positividade ≥ 50% de PD-L1 TC, de acordo com a rotulagem do
produto terapêutico aprovado.
Nota: A análise de imagem uPath PD-L1 (SP263) para o NSCLC
é uma metodologia auxiliar assistida por computador que visa
ajudar na aquisição e medição de imagens a partir de lâminas
de microscópio de espécimes de tecido corados IHQ, com vista a
verificar a presença da proteína PD-L1. Para assegurar a validade
das pontuações da análise de imagem, o patologista tem a
responsabilidade de verificar a respetiva conformidade utilizando
controlos apropriados, conforme é especificado na folha de
métodos do VENTANA PD-L1 (SP263) Assay (P/N 741-4905).
Este algoritmo destina-se ao diagnóstico in vitro (IVD).
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 3

Significado clínico
Há várias décadas que o cancro do pulmão é o cancro mais
comum a nível mundial, continuando a ser a principal causa de
morte por cancro em todo o mundo. Estima-se que represente
12,9% de todos os novos casos de cancro, sendo responsável
por aproximadamente 1,76milhões de mortes anuais em todo
o mundo, ou por aproximadamente uma em cada cinco mortes
relacionadas com cancro. O NSCLC é o tipo mais frequente de
cancro do pulmão, representando aproximadamente 85% de
todos os casos de cancro do pulmão. A maioria dos pacientes
com NSCLC apresenta um quadro de doença localmente
avançada, inoperável (Estádio IIIB) ou doença metastática
(Estádio IV), e nenhuma delas possui atualmente quaisquer
opções de tratamento curativo. A taxa de sobrevivência relativa
aos 5 anos para o NSCLC diagnosticado em estádios avançados
é de 4,7%.
O ligando 1 de morte celular programada (PD-L1) é uma proteína
transmembranar expressa em células T ativadas que regula
negativamente a resposta imunitária, ligando-se aos seus dois
recetores inibitórios de morte programada-1 (PD-1) e B7-1
(CD80). A ligação de PD-L1 à PD-1 inibe a proliferação de
células T, a produção de citocina e a atividade citolítica, levando à
inativação funcional ou à exaustão das células T.
A PD-L1 também se liga ao CD80 em células com presença de
antigénios e células T ativadas, mediando a regulação negativa
das respostas imunitárias, incluindo a inibição da ativação das
células T e a produção de citocina. A expressão de PD-L1 é
observada em células do sistema imunitário e em TC. Tem sido
reportado que a expressão aberrante de PD-L1 em TC impede a
imunidade antitumoral, resultando em evasão imunitária.
O aparecimento de imunoterapias que interrompem a via
PD-L1/PD-1 tem demonstrado melhorar as taxas de sobrevivência
nos pacientes com diagnóstico de NSCLC. No entanto, o
prognóstico do tratamento depende do nível de expressão
de PD-L1 e, por conseguinte, requer a quantificação de PD-L1
com coloração imunohistoquímica.
A imunohistoquímica pode ser utilizada para detetar antigénios
específicos presentes em amostras de tecido, sendo, por isso, uma
ferramenta eficaz para os patologistas, em termos de diagnóstico
e prognóstico de carcinomas. O VENTANA PD-L1 (SP 263)
Assay é um anticorpo monoclonal de coelho que se destina a ser
utilizado no laboratório para deteção semiquantitativa da proteína
PD-L1 em secções de tecido normal e neoplásico FFPE.
Uma vantagem das preparações histológicas de tecido é o
facto de o tecido intacto permitir que a morfologia auxilie na
interpretação da positividade de PD-L1 da amostra de paciente.
Todos os testes histológicos devem ser interpretados por
um especialista em morfologia e/ou patologia do NSCLC. Os
resultados devem ser complementados por estudos morfológicos
e controlos adequados, e utilizados em conjunto com outros
dados clínicos e laboratoriais.
4 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

Princípios de teste
O uPath enterprise software com o algoritmo de análise de imagem
uPath PD-L1(SP263) para o NSCLC recorre a técnicas de análise
de imagem para obter uma pontuação de positividade de TC.
O algoritmo de análise de imagem uPath PD-L1(SP263) para o
NSCLC utiliza parâmetros predefinidos para pontuar as imagens
de tecido coradas com o VENTANA PD-L1 (SP263) Assay.
Passos envolvidos na análise de imagem:
• Identificação de espaço em branco e exclusão automática da
análise.
• Deteção de células em toda a imagem.
• Classificação das células como TC ou células de outro tipo.
• Identificação de TC coradas versus não coradas.
• Cálculo da pontuação de positividade de TC dividindo o
número de TC coradas pelo número total de TC, de acordo
com a folha de métodos do VENTANA PD-L1 (SP263) Assay.
De que forma é que o algoritmo de análise de imagem
uPath PD-L1(SP263) para o NSCLC identifica as TC e como
é calculada a pontuação:
• O algoritmo de análise de imagem uPath PD-L1(SP263)
para o NSCLC identifica as TC utilizando características
morfológicas, cor, intensidade e dimensão.
• As TC identificadas são classificadas como coradas utilizando
a membrana detetada e limiares predefinidos, em linha com
a folha de métodos do VENTANA PD-L1 (SP263) Assay.
• Para calcular a percentagem de TC coradas, o número de TC
coradas é dividido pelo número total de TC, de acordo com a
folha de métodos do VENTANA PD-L1 (SP263) Assay.
Determinação de espaço em branco do algoritmo de
análise de imagem uPath PD-L1(SP263) para o NSCLC:
• O algoritmo vai excluir automaticamente o espaço em branco.
Artefactos como sujidade, riscos ou tinta podem não ser
excluídos automaticamente.
• O utilizador pode rever as áreas que foram determinadas
pelo algoritmo como espaço em branco utilizando uma
sobreposição de cor falsa; consulte a secção "Análise de
imagem de PD-L1: sobreposição de cor falsa".
Critérios do algoritmo de análise de imagem uPath
PD-L1(SP263) para o NSCLC para geração do resultado da
pontuação de positividade de TC:
• O algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC vai reportar como resultado uma pontuação que terá
um dígito na casa decimal, por ex. "4,8%".
• Não se recomenda que esta pontuação seja arredondada
por excesso ou por defeito. Por exemplo, "4,8%" não deve ser
arredondado por excesso para 5%.
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 5

O algoritmo de análise de imagem uPath PD-L1(SP263) para o
NSCLC foi concebido para funcionar com o VENTANA PD-L1
(SP263) Assay. A qualidade dos testes depende da qualidade e da
exatidão da imagem obtida da lâmina corada IHQ e da imagem
subsequente que é analisada.
O patologista deve validar a execução da coloração do VENTANA
PD-L1 (SP263) Assay através de um exame das lâminas de
controlo de PD-L1, utilizando um microscópio manual, a fim de
verificar se foram obtidos os resultados previstos, antes de as
imagens das lâminas serem aceites no uPath enterprise software
para análise.
Utilize as recomendações do fabricante do VENTANA PD-L1
(SP263) Assay, incluindo todos os materiais de controlo da
qualidade positivos e negativos em cada execução de coloração.
Se as lâminas de controlo não forem aceitáveis após exame com
microscópio manual, deve fazer novamente a coloração de tecido
que tenha resultados aceitáveis.
O patologista tem de seguir as recomendações de interpretação
do VENTANA PD-L1 (SP263) Assay.
Consulte a folha de métodos (P/N 1014258PT) e o guia de
interpretação (P/N 1015317EN) do VENTANA PD-L1 (SP263)
Assay (disponível em www.ventana.com).
O algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC foi concebido para ser utilizado por um patologista
qualificado, em conjunto com um exame histológico, a informação
clínica relevante e os controlos adequados. Não foi concebido
como ferramenta autónoma e requer intervenção humana
competente durante todo o processo de análise.
O algoritmo de análise de imagem uPath PD-L1(SP263) para o
NSCLC pode gerar pontuações incorretas se as imagens capturadas
tiverem uma coloração anormal (nuclear, citoplasma, etc.).
O algoritmo de análise de imagem uPath PD-L1(SP263)
para o NSCLC irá rejeitar núcleos tumorais alongados,
independentemente do formato geral da célula. Por esta razão,
tumores que contenham grandes quantidades de células com
núcleos alongados poderão ter de ser avaliados manualmente.
Limitações
O algoritmo de análise de imagem uPath PD-L1(SP263) para o
NSCLC foi estudado, desenvolvido e validado em amostras de
tecido de NSCLC.
O algoritmo de análise de imagem uPath PD-L1(SP263) para o
NSCLC não foi testado, ou a sua segurança e eficácia não foram
validadas, quando utilizado com um computador pessoal (PC) a
partir de casa.
Está indicada a utilização do algoritmo de análise de imagem
uPath PD-L1(SP263) como auxiliar na identificação dos pacientes
com NSCLC que vão efetuar tratamento com terapêuticas com o
valor limite de positividade de 50% de PD-L1 TC, de acordo com a
rotulagem do produto terapêutico aprovado.
O algoritmo de análise de imagem uPath PD-L1(SP263) para
o NSCLC pode identificar incorretamente as células devido à
presença de coloração citoplasmática e/ou membranosa fraca,
coloração forte das células do sistema imunitário sobreposta
com coloração das células tumorais, quando exista inflamação
significativa misturada, TC com ligeira coloração citoplasmática e
coloração sem alvo. Daqui pode resultar a identificação incorreta
das TC com não TC, e das não TC como TC positivas.
Embora seja necessária a exclusão de macrófagos da região
de análise, nem sempre é possível excluir todos os macrófagos.
Em resultado disto, a pontuação gerada pelo algoritmo de
análise de imagem uPath PD-L1(SP263) para o NSCLC pode ser
influenciada pelos macrófagos na ROI que está a ser analisada.
Isto é crucial quando a pontuação de um paciente está próxima
do valor limite de 50%.
A coloração citoplasmática é geralmente difusa, verificando-se
em alguns casos uma granulosidade fina. Casos raros mostraram
uma coloração dos corpos perinuclear semelhante a pontos,
com intensidade variável. A percentagem total de intensidades
de sinais das membranas tumorais é estimada visualmente e
usada para gerar o nível de expressão de PD-L1. A coloração
citoplasmática das células tumorais não é considerada para
determinar a expressão de PD-L1. Utiliza-se um anticorpo de
controlo negativo com o mesmo isótopo para avaliar a presença
da coloração de fundo em amostras de teste e estabelecer uma
referência de intensidade da coloração.
6 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

Proteção de dados
O acesso não autorizado ao equipamento ou um software
malicioso pode ter como resultado a perda de dados ou a
indisponibilidade do equipamento.
Para evitar que o equipamento seja infetado por software
malicioso ou que ocorra um acesso não autorizado e a utilização
indevida do equipamento, é essencial seguir as recomendações
que se seguem:
• Não instale nem execute qualquer outro software no
equipamento.
• Certifique-se de que os outros computadores e serviços na
rede estão devidamente protegidos e seguros contra software
malicioso e acesso não autorizado. Por exemplo, o sistema
de informações laboratoriais (LIS), a partilha de arquivo, a
partilha de cópias de segurança ou a assistência.
• Os clientes são responsáveis pela segurança da sua rede
de área local, especialmente pela proteção da rede contra
software e ataques maliciosos. Esta proteção pode incluir a
implementação de medidas como, por exemplo, uma firewall,
para proteger o dispositivo contra redes não controladas. Esta
proteção pode igualmente incluir medidas que assegurem que
a rede conectada não contém código malicioso.
• Restrinja o acesso físico ao equipamento e a toda a
infraestrutura de TI associada (computadores, cabos,
equipamento de rede e assim sucessivamente).
• Certifique-se de que os ficheiros de arquivo e cópia de
segurança do equipamento estão protegidos contra qualquer
acesso não autorizado e contra desastres. Esta lista inclui a
localização de armazenamento remoto, sites de descoberta
de desastres e a transferência segura de ficheiros de cópia de
segurança.
• Se possível, utilize uma firewall para restringir o tráfego na
rede.
• Podem ser utilizadas pens USB para realizar e restaurar
diferentes tipos de cópias de segurança. A utilização incorreta
destas pens USB pode ter como resultado a perda de dados ou
uma avaria do equipamento.
• Utilize apenas pens USB que tenham sido testadas e instaladas
pela assistência técnica da Roche local.
• Só pode ser utilizado um dispositivo USB de cada vez. Antes
de introduzir uma pen USB, verifique se não está a ser utilizado
mais nenhum dispositivo USB.
• Antes de remover uma pen USB, escolha o botão Eject (Ejetar)
no Windows.
• A configuração predefinida do sistema operativo (SO)
fornecida com o servidor não deve ser alterada, porque se for
isso terá implicações nas configurações do SO com proteção.
• Para impedir que o uPath enterprise software seja infetado
por um vírus, utilize a pen USB única e exclusivamente nesse
equipamento. Não armazene outros dados nesta pen USB.
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 7

Fluxo de trabalho para utilização do algoritmo de análise de imagem uPath
PD-L1 para o NSCLC
Materiais necessários
• uPath enterprise software
• Algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC
• Lâminas de tecido de NSCLC coradas com o VENTANA PD-L1 (SP263) Assay (utilizando o OptiView DAB IHC Detection Kit) no
equipamento BenchMark ULTRA
• Digitalizador de lâminas VENTANA DP 200
Fluxo de trabalho
1. Uma amostra de tecido de NSCLC depositada numa lâmina é corada com o VENTANA PD-L1 (SP263) Assay utilizando um
equipamento BenchMark ULTRA.
2. É efetuada a aquisição de imagem (digitalização de toda a lâmina) com o digitalizador de lâminas VENTANA DP 200 utilizando uma
ampliação de 20x num plano z.
3. Depois de adquiridas, as imagens digitais são transferidas do computador associado ao digitalizador de lâminas VENTANA DP 200
para o sistema de gestão de imagens (Image Management System, IMS) num servidor centralizado.
4. A seguir à transferência para o servidor, é criado um caso no uPath enterprise software. A criação do caso pode ocorrer
automaticamente, através de comunicação com o sistema de informações laboratoriais (LIS), utilizando as informações de
identificação (i.e., tipo de tecido e anticorpo primário) contidas na etiqueta de código de barras da lâmina, ou através de introdução
manual no uPath enterprise software (consulte o Manual do utilizador do uPath enterprise software (P/N 1018943PT)).
5. Se o algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC estiver instalado (tem de estar instalado num servidor
separado do uPath enterprise software e do IMS) e for aceite uma imagem ampliada 20x com a coloração e o tipo de tecido
apropriado, o uPath enterprise software aciona automaticamente a análise da lâmina completa (Whole Slide Analysis, WSA).
6. A WSA analisa automaticamente toda a imagem digitalizada.
7. Uma vez concluída a WSA, o patologista é notificado dentro do uPath enterprise software através da mensagem "analysis is
complete" (a análise está concluída). O patologista poderá então selecionar ROI específicas e atribuir-lhes uma pontuação. O
patologista pode selecionar uma só ROI de qualquer dimensão, ou várias ROI. Se selecionar várias ROI, é atribuída uma pontuação
agregada, mas também é atribuída uma pontuação individual a cada ROI.
Coloração
• A preparação e a coloração do tecido devem ser feitas de acordo com as recomendações da folha de métodos do VENTANA PD-L1
(SP263) Assay.
• Todos os controlos adequados deverão ser revistos e as lâminas deverão ser coradas de novo se a coloração não cumprir as linhas
de orientação enunciadas na folha de métodos do VENTANA PD-L1 (SP263) Assay.
• O algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC requer que seja utilizado o VENTANA PD-L1 (SP263) Assay,
bem como quaisquer materiais ou abastecimentos adicionais referidos na folha de métodos do VENTANA PD-L1 (SP263) Assay,
para efetuar a coloração dos tecidos antes da análise. O VENTANA PD-L1 (SP263) Assay deteta a proteína PD-L1 em tecido de
NSCLC FFPE corado com o OptiView DAB IHC Detection Kit num equipamento BenchMark ULTRA. Embora o VENTANA PD-L1
(SP263) Assay detete a proteína PD-L1 em tecido de NSCLC FFPE corado nos equipamentos BenchMark ULTRA, o algoritmo
de análise de imagem uPath PD-L1(SP263) para o NSCLC foi validado utilizando o OptiView DAB IHC Detection Kit corado no
equipamento BenchMark ULTRA.
Captura de imagens
Para digitalizar as lâminas, é necessário um digitalizador de lâminas VENTANA DP 200. As imagens têm de ser digitalizadas com uma
ampliação de 20x. Se houver secções grandes da imagem que estejam desfocadas, recomenda-se que as lâminas sejam digitalizadas
novamente. Para mais informações sobre o processo de digitalização, consulte o Manual do Utilizador do digitalizador de lâminas
VENTANA DP 200 (PN 1017149PT).
8 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

Navegação geral: uPath enterprise software
O uPath enterprise software destina-se a ser personalizado em função das necessidades individuais e da instituição, incluindo, sem
limitações, a configuração de relatórios e a interface de utilizador. Este Guia do algoritmo irá concentrar-se nas ferramentas necessárias
à utilização apenas do algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC. Para mais informações sobre o uPath
enterprise software, consulte o Manual do utilizador do uPath enterprise software.
Fluxo de trabalho do patologista
Abrir um caso
Imagens do tecido de NSCLC corado com o VENTANA PD-L1 (SP263) Assay podem ser acedidas fazendo duplo clique num caso ou
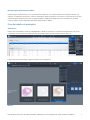
selecionando um caso e premindo o separador Viewer (Visualização) dentro do uPath enterprise software (Figura 1).
É apresentado um ecrã com todas as imagens associadas a um caso (Figura 2).
Figura 2
Figura 1
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 9

Depois de uma lâmina corada com o VENTANA PD-L1 (SP263) Assay ser digitalizada num digitalizador de lâminas VENTANA DP
200 com uma ampliação de 20x, a imagem é importada para o uPath enterprise software e associada a um caso. O algoritmo de
análise de imagem uPath PD-L1(SP263) para o NSCLC aciona automaticamente a WSA. O tempo necessário para concluir o passo
de pré-cálculo da WSA depende das especificações do servidor, do tamanho da imagem e do número de imagens que estiver na fila.
Se for apresentada a mensagem "waiting to start auto-analysis" (a aguardar para iniciar a análise automática), isso significa que as
imagens estão na fila e ainda não foram analisadas. Se for apresentada a mensagem "analyzing" (a analisar), isso significa que está a
ser executada a WSA (Figuras 3 e 4). Depois de a imagem ter sido completamente analisada via WSA no uPath enterprise software, é
apresentada a mensagem "analysis successful" (análise bem-sucedida) por baixo da imagem da lâmina no Viewer (Visualização) do
uPath enterprise software (Figura 5). Não é possível pontuar as imagens se a WSA não tiver sido concluída com êxito.
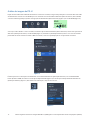
Desenhar a(s) ROI do total do tumor: selecionar a área do tumor
Utilize o botão da ferramenta Freehand (Mão livre) no menu pendente (Figura 6) para selecionar a(s) área(s) do tumor na imagem da
lâmina corada IHQ que vai ser analisada. A Figura 7 ilustra uma imagem com uma única ROI desenhada. É possível desenhar mais ROI.
Cada área selecionada fará com que seja apresentada uma ROI no Slide Panel (Painel de lâminas) (Figure 8).
Figura 6 Figura 7
Figura 8
Figura 5
Figura 4
Figura 3
10 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

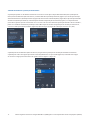
Desenhar a(s) ROI do total do tumor: área de exclusão
Ao desenhar as ROI, pode ser necessário excluir algumas áreas. Na secção abaixo relativa às características de coloração, iremos
abordar áreas específicas que devem ser evitadas ou omitidas e apresentar exemplos. Utilize a ferramenta de exclusão Freehand
(Mão livre) do menu pendente Exclusion (Exclusão) (Figura 9) para excluir áreas específicas (Figura 10). Se houver grandes áreas da
imagem enevoadas ou desfocadas, digitalize novamente a lâmina.
As áreas excluídas não serão analisadas pelo algoritmo, e as TC coradas e não coradas dentro desta área serão excluídas da área
de análise total. Se a ROI já tiver sido analisada e for utilizada uma exclusão, a ROI terá de ser reanalisada e a sobreposição será
devidamente atualizada.
Desenhar um grande número de exclusões, especialmente exclusões Freehand (Mão livre) complicadas, pode ser moroso e ter impacto
na eficiência do fluxo de trabalho, tendo apenas um impacto marginal da pontuação final. Se um caso necessitar de um grande número
de exclusões, o patologista deve fazer o seguinte:
• Desenhar várias ROI e excluir porções de tecido que considere não pontuáveis utilizando o mínimo possível a ferramenta Exclusion
(Exclusão).
• Limitar as exclusões e substituir manualmente a pontuação por outra pontuação.
Desenhar a(s) ROI do total do tumor: eliminação
Se uma ROI do total do tumor selecionada não for de qualidade ótima, pode ser eliminada. Selecione a ROI do total do tumor clicando no
centro da ROI na imagem e depois clicando no botão Delete (Eliminar) dentro do Slide Panel (Painel de lâminas) (Figura 11) ou dentro
da imagem da lâmina, próximo da ROI (Figura 12). Será apresentada uma janela de confirmação. Selecione Confirm (Confirmar) para
eliminar a ROI selecionada. Selecione Cancel (Cancelar) para manter a ROI.
Figura 10Figura 9
Figura 11 Figura 12
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 11

Depois de desenhadas todas as ROI do total do tumor e/ou áreas de exclusão, a imagem está pronta para ser analisada. Selecione a ROI
do total do tumor clicando no centro da ROI que vai ser analisada ou clicando na ROI no Slide Panel (Painel de lâminas). Para cada ROI,
selecione o botão Image Analysis (Análise de imagem) dentro do Slide Panel (Painel de lâminas) (Figura 13) ou ao lado da ROI (Figura 14).
Assim que a análise de PD-L1 estiver concluída, o resultado é apresentado no Slide Panel (Painel de lâminas) em dois sítios: por baixo da
Slide Score (Pontuação da lâmina) e ao lado das ROI (Figura 15). A Slide Score (Pontuação da lâmina) baseia-se na soma do estado de
positividade das células tumorais em todas as ROI selecionadas, e será esta a pontuação que vai ser apresentada no relatório.
Análise de imagem de PD-L1
Figura 15
É também possível ver informações mais detalhadas no ecrã detalhado Slide Score (Pontuação da lâmina) e no ecrã detalhado ROI
Details (Detalhes da ROI) clicando no ícone de apresentação detalhada (Figura 16). É aberta a apresentação detalhada da Slide Score
(Pontuação da lâmina) (Figura 17). Para ocultar estas informações, clique no mesmo ícone.
Figura 17
Figura 16
Figura 13 Figura 14
12 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

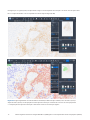
Análise de imagem de PD-L1: sobreposição de cor falsa
Depois de ter sido premido o botão ROI(s) Analysis (Análise de ROI) e de o tecido ter sido analisado, é aplicada uma sobreposição de
cor sobre a ROI. Na imagem abaixo (Figura 18), os círculos vermelhos representam células que se determinou terem coloração positiva
para PD-L1 e os círculos azuis representam células que se determinou serem negativas para PD-L1. Quando se agarra na imagem
(clicando com o botão esquerdo do rato e movendo a imagem) a sobreposição desaparece (Figura 19). Quando se solta o botão do rato,
a sobreposição reaparece (Figura 18).
Figura 18
Figura 19
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 13

Substituir manualmente a pontuação de uma lâmina
As pontuações podem ser substituídas manualmente clicando no ícone de apresentação detalhada de Slide Score (Pontuação da
lâmina) dentro do Slide Panel (Painel de lâminas), ao lado da Slide Score (Pontuação da lâmina) (Figura 16). É aberta a apresentação
detalhada da Slide Score (Pontuação da lâmina) (Figura 20). Selecionando o botão Edit (Editar) (Figura 20) na apresentação detalhada
da Slide Score (Pontuação da lâmina), o utilizador pode introduzir uma pontuação manualmente (Figura 21). O campo Comments
(Comentários) permite que notas relacionadas com o caso e/ou com a decisão substituam a pontuação automática. No caso da PD-L1,
podem ser introduzidas manualmente pontuações de 0 a 100%. Depois de introduzir uma pontuação de substituição manualmente,
selecione a notificação Confirm (Confirmar) (Figura 22). Quando surgir a mensagem de confirmação, selecione "Yes" (Sim).
A pontuação dentro do Slide Panel (Painel de lâminas) irá agora refletir a pontuação de substituição introduzida manualmente.
A pontuação de análise de imagem apresentada ao lado da(s) ROI deixa de ser apresentada (Figura 23). O utilizador terá a opção
de reanalisar a imagem premindo o botão com o gráfico de barras (Figura 13, Figura 14).
Figura 21 Figura 22
Figura 20
Figura 23
14 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)

Consulte a folha de métodos e o Guia de interpretação do VENTANA PD-L1 (SP263) Assay.
Avaliação de PD-L1
As células neoplásicas de NSCLC marcadas com o VENTANA PD-L1 (SP263) Assay são avaliadas quanto à positividade percentual
das
TC com coloração das membranas em qualquer intensidade. A coloração imunohistoquímica no NSCLC é membranosa e/ou
citoplasmática
e pode ser expressa homogénea ou heterogeneamente através do neoplasma. A coloração citoplasmática de células tumorais não
é contabilizada na pontuação de positividade de TC. A coloração membranar pode ter um padrão descontínuo, circunferencial ou
basolateral. Utiliza-se um anticorpo de controlo negativo com o mesmo isótopo para avaliar a presença de coloração de fundo em
amostras de teste.
Avaliação da coloração do algoritmo PD-L1
O patologista que utilizar o algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC deve estar familiarizado com a
pontuação manual do VENTANA PD-L1 (SP263) Assay. O patologista deve utilizar a ferramenta Freehand (Mão livre) para contornar
a área total do tumor. O patologista deve utilizar como referência a lâmina de controlo negativo e H&E associada antes de utilizar o
algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC. Quando selecionar as áreas que devem ser analisadas, tenha em
consideração as limitações descritas nas secções Limitações e Áreas a evitar. Se o patologista discordar da pontuação fornecida pelo
algoritmo de análise de imagem uPath PD-L1(SP263) para o NSCLC, deve substituir manualmente essa pontuação.
Os casos não avaliáveis são, entre outros, casos com tumor viável insuficiente, morfologia inaceitável e fundo interferente. Os casos de
NSCLC com TC viáveis suficientes (conforme determinado pela pontuação do patologista) e sem fundo interferente na lâmina corada
IHQ de PD-L1 são aceitáveis para avaliação.
Características de coloração
Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC) 15

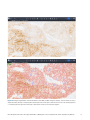
Figura 24: Imagens digitalizadas de tecido de NSCLC corado IHQ no uPath enterprise software; antes da análise (acima) e
depois da análise (abaixo). A sobreposição vermelha representa células que se determinou serem TC com coloração positiva
e a sobreposição azul representa células que se determinou serem TC com coloração negativa.
Nas figuras que se seguem (24-25) são apresentadas imagens com vários padrões de coloração e com vários níveis de expressão de
PD-L1. A expressão de PD-L1 nas TC é reportada sob a forma de percentagem (0-100).
16 Guia do algoritmo de análise de imagem uPath PD-L1(SP263) para o cancro do pulmão de células não pequenas (NSCLC)
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
A página está carregando...
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Manual do usuário
- Tipo
- Manual do usuário
Artigos relacionados
-
Roche VENTANA PD-L1 (SP263) Assay, Universal, CE IVD, Predictive, NSCLC Interpretation Guide
-
Roche uPath IVD Manual do usuário
-
Roche VENTANA anti-ALK D5F3 Primary Antibody Manual do usuário
-
Roche VENTANA PD-L1 (SP142) Assay Interpretation Guide
-
Roche uPath HER2 (4B5) image analysis for Breast Algorithm Manual do usuário
-
Roche BenchMark XT/LT Interpretation Guide






































